
INTRODUCCIÓN
El grupo continúa el exhaustivo estudio de la epigenética que Manel Esteller ha llevado a cabo como investigador principal durante toda su carrera científica. En la actualidad, las investigaciones se centran en el establecimiento de mapas epigenómicos y epitranscriptómicos para células normales y transformadas, en el estudio de las interacciones entre las modificaciones epigenéticas y los ARN no codificantes y en el desarrollo de nuevos fármacos epigenéticos para el tratamiento del cáncer.
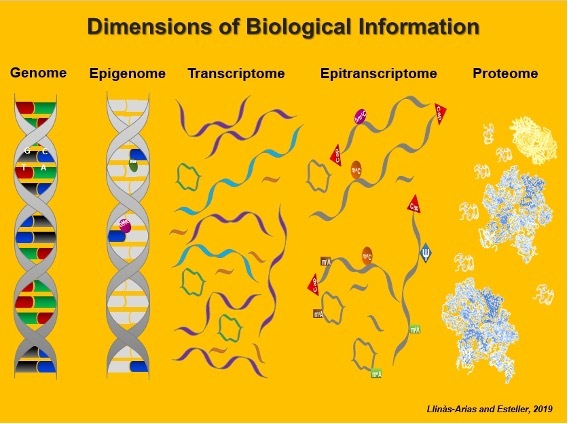
NUESTRA INVESTIGACIÓN
Nuestro laboratorio es uno de los responsables de establecer que la interrupción epigenética de la transcripción del ARNm, en particular los patrones de metilación del ADN y de modificación de histonas, contribuye al inicio y la progresión de los tumores en humanos (revisado en Esteller, N Engl J Med 2008; Heyn y Esteller, Nat Rev Genet 2012; Berdasco y Esteller, Nat Rev Genet 2019); Davalos y Esteller, CA Cancer J Clin 2023).
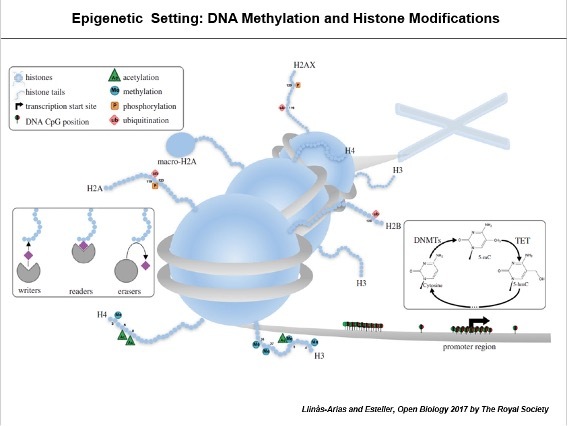
También se ha identificado que los micro-ARN (ARN pequeños no codificantes que regulan la expresión génica mediante emparejamiento de bases específico de secuencias en dianas de ARNm) desempeñan asimismo un papel clave en la biología celular y pueden afectar al desarrollo del cáncer. En este contexto, llevamos a cabo la caracterización del primer micro-ARN sometido a silenciamiento asociado a metilación específico del cáncer (Lujambio et al., Cancer Res 2007), seguido de la caracterización de otros muchos micro-ARN alterados de forma similar (Lujambio et al., PNAS 2008; Davalos et al., Oncogene 2012).
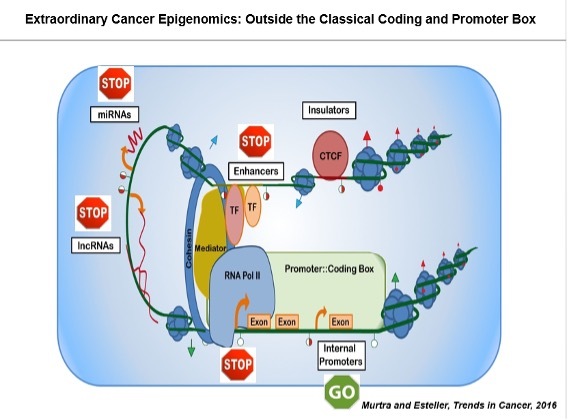
NUESTROS OBJETIVOS
Nuestro grupo está interesado desde hace tiempo en trasladar el uso del conocimiento de la epigenética logrado gracias a la investigación al uso de biomarcadores para predecir desenlaces clínicos y para evaluar nuevos fármacos que reviertan el distorsionado panorama epigenético (Berdasco y Esteller, Nature Review Genetics 2019). Así, por ejemplo, hemos usado marcadores epigenéticos para predecir la respuesta a terapias antitumorales tras la observación inicial de que la metilación del gen MGMT predecía la respuesta a agentes alquilantes en el glioma (Esteller et al., N Engl J Med 2000).
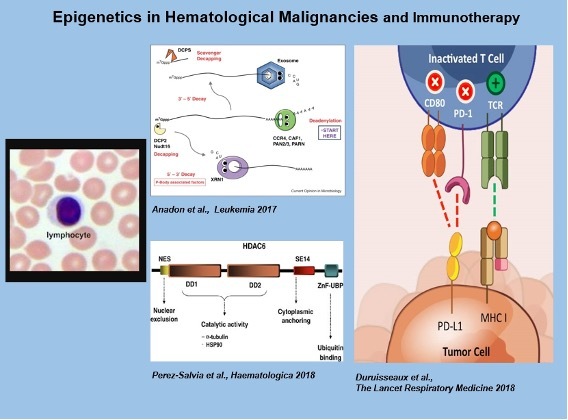
NUESTROS RETOS
Continuando con este aspecto traslacional de nuestro trabajo, también estamos interesados en el desarrollo y el estudio de nuevos fármacos epigenéticos dirigidos a escritores, lectores y borradores de la modificación de histonas y la metilación de ADN, y que podrían tener un efecto antineoplásico (Lara et al., Oncogene 2008; Zubia et al., Oncogene 2009; Huertas et al., Oncogene 2012; Perez-Salvia et al., Oncotarget 2017; Perez-Salvia et al., Haematologica 2018).
Equipo

Jefe de grupo senior

Investigador/a Asociado/da

Investigador/a Asociado/da

Investigador/a Asociado/da

Investigador/a Asociado/da

Investigador/a Postdoctoral
Investigador/a Postdoctoral

Investigador/a Postdoctoral

Investigador/a Postdoctoral

Investigador/a Postdoctoral

Investigador/a Postdoctoral

Investigador/a Postdoctoral

Investigador/a predoctoral

Investigador/a predoctoral
Investigador/a predoctoral

Investigador/a predoctoral

Investigador/a predoctoral

Investigador/a predoctoral

Investigador/a predoctoral

Investigador/a predoctoral

Investigador/a predoctoral

Técnico/a de laboratorio superior
Publicaciones seleccionadas
Ayudas vigentes
Ministerio de ciencia e innovación
COMBATIR NUEVAS ESTRATEGIAS COMBINATORIAS Y BIOMARCADORES DE RESPUESTA A INHIBIDORES DE HDAC6 PARA EL TRATAMIENTO DEL CÁNCER.
European academy of dermatology and venereology
SCIENCE-SS Single-cell Characterization of Epigenetic and Transcriptomic Dynamics in Sézary Syndrome Progression
Fundació "la caixa"
Solve-ALD Brain Inflammation in adrenoleukodystrophy: from drivers to treatments
Instituto de salud carlos iii
SEED-ALS Synergizing Efforts to Develop and Accelerate Breakthroughs in ALS Research
Gilead sciences s.l.
Identification of DLBCL key molecular features for CAR-T cell therapy
European commission
THRIVE TUMOUR-HOST INTERACTIONS IN LIVER CANCER OF CHILDHOOD AND ADULTS

Fundació "la caixa"
Solve-ALD Brain Inflammation in adrenoleukodystrophy: from drivers to treatments

Fundación científica de la asociación española contra el cáncer
PREDICT-Meso PRE-malignant Drivers Combined with Target-Drug validation in Mesothelioma

European commission
SENESCENCE2030 Targeting Cell Senescence to Prevent Age-Related Diseases
Deutsche josé carreras leukämie stiftung
scEpiCART Refinement of Epigenetic Factors to Improve CAR T Cell Therapeutic Efficacy
Fundación mutua madrileña
Identificación de determinantes de respuesta a quimioinmunoterapia en biopsia líquida, en pacientes con cáncer microcítico de pulmón.

Ministerio de ciencia e innovación
ASPIRE Neoadjuvant-adjuvant immunotherapy and cancer vaccines to improve survival in hepatocellular carcinoma
Instituto de salud carlos iii
CIBERONC Centro de Investigación Biomédica en Red Cáncer
Cll global research foundation
Gene Expression Regulation Study Mediated by the RNA-Binding Protein Musashi2 and the RNA Methyladenosine Transferase 3, as Therapeutic Targets in Chronic Lymphocytic Leukemia
Fundació "la caixa"
MyoClonal Somatic mutations and clonal hematopoiesis as predictors and drivers of heart failure progression

Ministerio de ciencia e innovación
Single cell analysis of clonal heterogeneity in myelodysplastic syndromes treated with azacitidine

European commission
IRB-TARGET IRB Barcelona International PhD programme: on TARGET for high-impact biomedicine

Fundación científica de la asociación española contra el cáncer
T-REX Transcriptional Role and Epigenetics of the X chromosome in leukemias
Agència de gestió d'ajuts universitaris i de recerca
Methylation Maps for Precision Hematology: Decoding Epigenetic Signatures of MDS Progression and Venetoclax Resistance in AML
Agència de gestió d'ajuts universitaris i de recerca
Overcoming Cancer Immunotherapy Resistance

Ministerio de ciencia e innovación
USO DE APROXIMACIONES DE CELULA UNICA PARA DESCIFRAR LA EPIGENOMICA DEL CANCER Y LAS EPIDROGAS

Ministerio de universidades
Estudios de epigenética del cáncer a nivel de célula única
